近日,我校生物信息团队杨庆勇课题组联合新加坡国立大学研究成果以“Calling large indels in 1047 Arabidopsis with IndelEnsembler”为题在“Nucleic Acids Research”发表。研究开发出高性能的大片段插入删除变异(InDel)鉴定工具‘IndelEnsembler’,使大片段InDel鉴定准确性由45%提高到84%。
遗传变异是指一个群体中个体之间DNA序列的差异,主要包括点突变(SNP/单核苷酸多态性)、小片段InDel和结构变异(包括大片段InDel、染色体倒位、易位和拷贝数变异)。SNP被广泛应用于群体遗传学研究和疾病相关基因的研究。然后越来越多的研究表明,SNP并不能解释所有的表型差异,在人类中有超过1000种疾病由结构变异导致。
据介绍,大量的遗传学和分子生物学证据表明大片段InDel在解释影响一系列重要农作物的表型变异中起主要作用。例如,黄瓜基因组上一个大片段DNA序列拷贝数变化可以决定黄瓜的性别,包含该变异的黄瓜产量约为普通黄瓜的15倍。蟠桃因其果形独特,味甜多汁而受到人们喜爱,研究发现蟠桃基因组中含有一个1.67 Mb的倒位,而普通桃中则没有。但是,目前鉴定到的表型相关的大片段InDel的数量远远小于SNP,其主要原因是现有的方法不能精确的鉴定基因组上的大片段InDel。该研究通过整合四个已发表的方法开发出高性能的大片段InDel鉴定工具IndelEnsembler,并将其应用于由1047个拟南芥品系构成的自然群体中,通过全基因组关联分析(GWAS)鉴定到与重要性状相关的、新的大片段InDel。
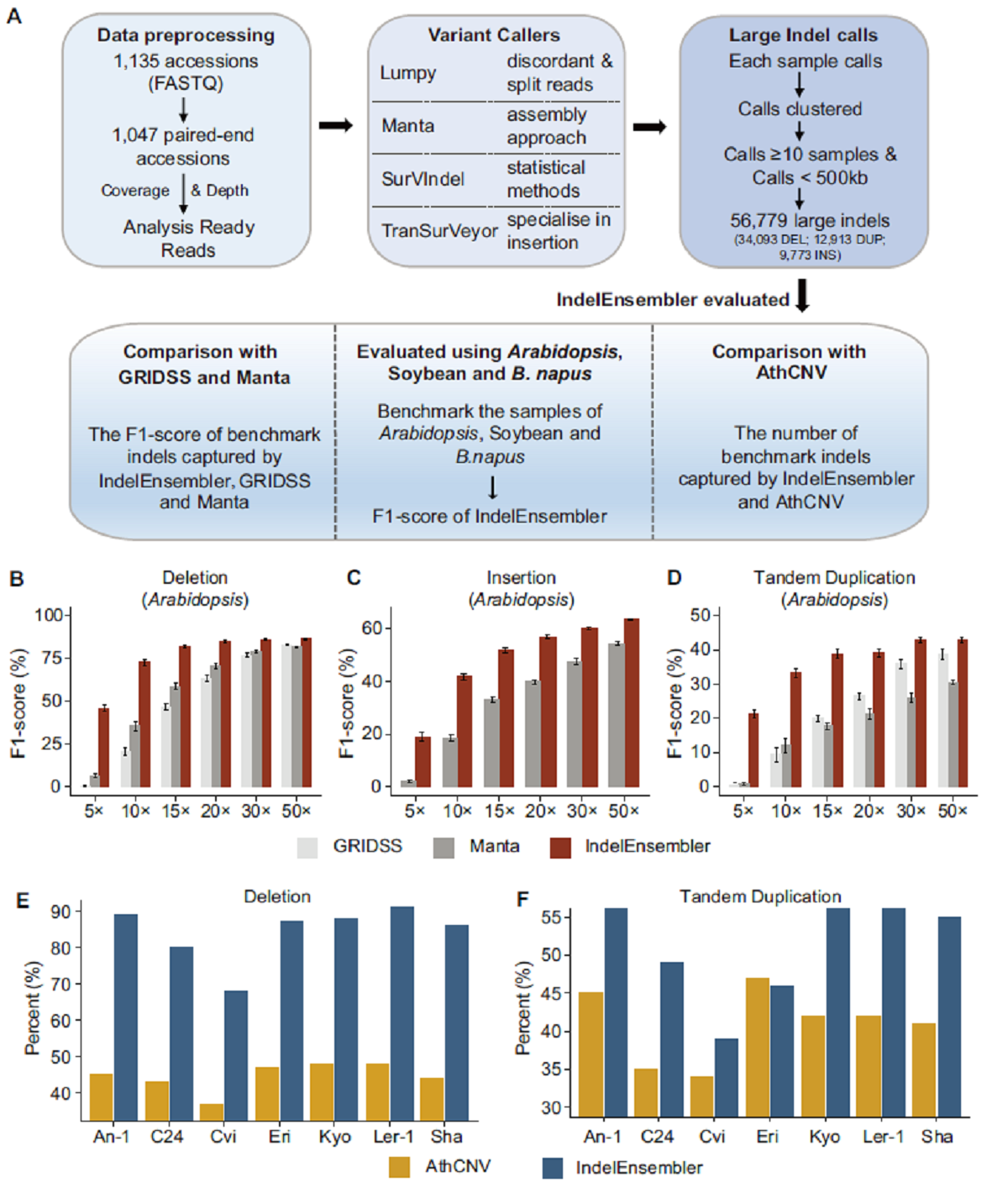
相比于目前鉴定InDel性能最佳的两个软件GRIDSS和Manta,IndelEnsembler在不同物种及不同测序深度下均有很好的性能。相比于拟南芥中已开发的工具AthCNV,IndelEnsembler在鉴定缺失变异(DEL)和重复变异(DUP)时准确性分别提高一倍和30%。
大片段InDel在拟南芥基因组上分布不均匀且与转座子的分布呈现极显著正相关,80-96%的染色体着丝粒区域被InDel覆盖。大片段InDel倾向于分布在基因间区以及非编码基因上。拟南芥基因组上有13,102个基因受到大片段InDel的影响,但是这些基因主要是未知功能的基因,说明大片段InDel影响的基因大部分为非核心基因。现有研究表明,非核心基因与水稻、玉米等重要农作物的适应性、品质和驯化等性状密切相关。此外,大片段InDel影响的基因更多的分布在基因组上的串联重复区域,说明基因组上的串联重复区域为不稳定区域会积累更多的变异。
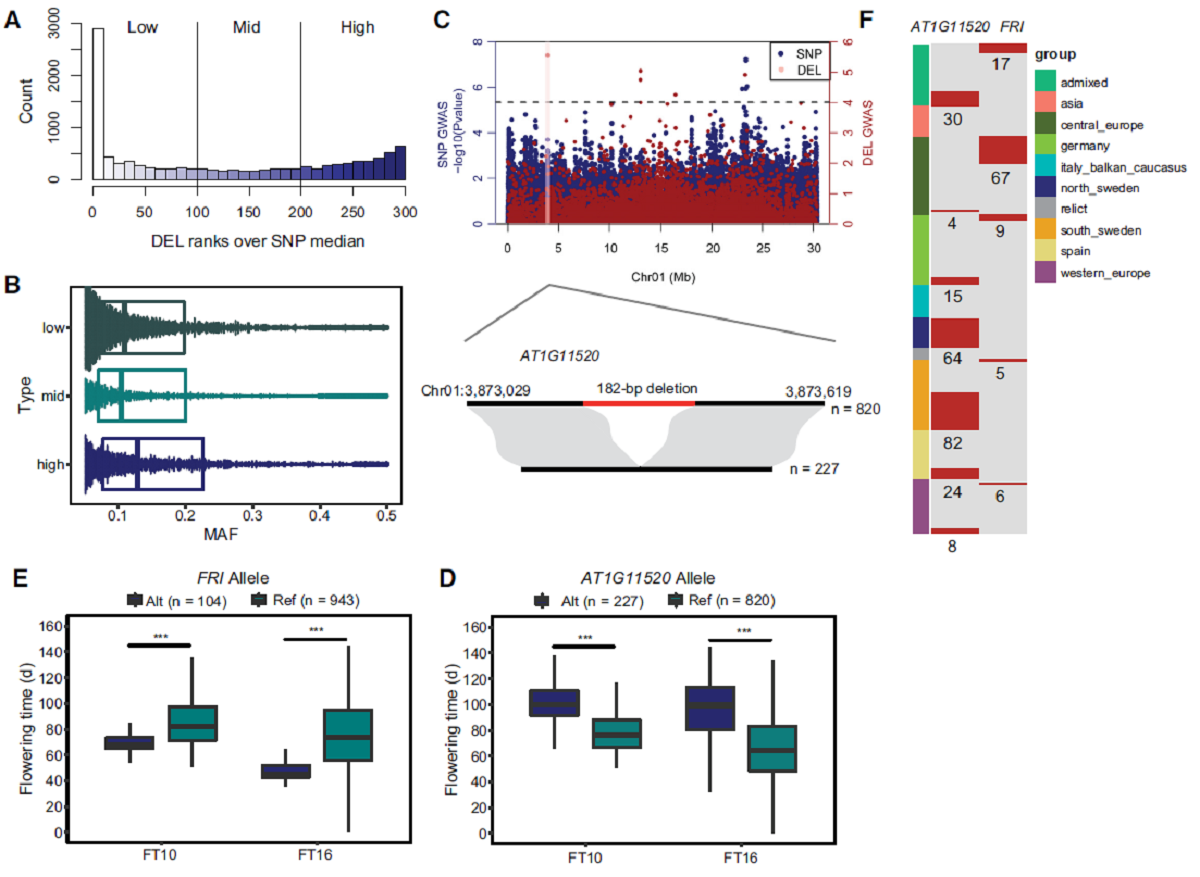
有48.91%的DEL与周围的SNP具有低连锁不平衡(LD),说明IndelEnsembler鉴定到了大量新的基因组变异,为挖掘影响拟南芥性状相关的基因组变异提供了重要资源。该研究鉴定到的一段377 bp的DEL造成开花期相关基因FRI第一个外显子上65 bp的缺失,含有该缺失的材料开花期提前。另外该研究还鉴定到AT1G11520基因上的一段182 bp缺失,含有该缺失的材料开花期推迟。值得注意的是,上述两段缺失不能在同一个材料中出现而且所有北瑞典地区的材料AT1G11520基因上均含有该182 bp的缺失。这些结果说明拟南芥中仍然存在不少未知的基因组变异,IndelEnsembler鉴定到的大片段InDel可以作为拟南芥中表型相关的基因组变异数据集的一个补充。
据悉我校信息学院博士研究生刘东旭和新加坡国立大学Ramesh Rajaby博士为该论文共同第一作者。
