近日,我校信息学院生物统计团队胡学海教授课题组研发出一款针对植物转录因子结合位点预测的工具及其docker镜像,相关研究成果发表在国际生物信息学领域学术期刊Bioinformatics上。
转录因子结合位点(TFBS)是顺式调控元件的基本组成部分,在基因表达的精确调控中起重要作用。TFBS核心基序内的非编码变异可能会显著改变其结合亲和力,这可能是解释遗传变异如何影响复杂性状的生物学机制。植物中转录因子结合位点实验数据的缺乏,以及植物TFs的独立进化特性都使得鉴定植物TFBS的计算方法落后于相关的人类研究。本研究首先使用深度卷积神经网络(DeepCNN)在基于可用的拟南芥Dap-seq数据集建立了265个拟南芥TFBS的预测模型,并且将其迁移用于预测其他植物的同源TF中。
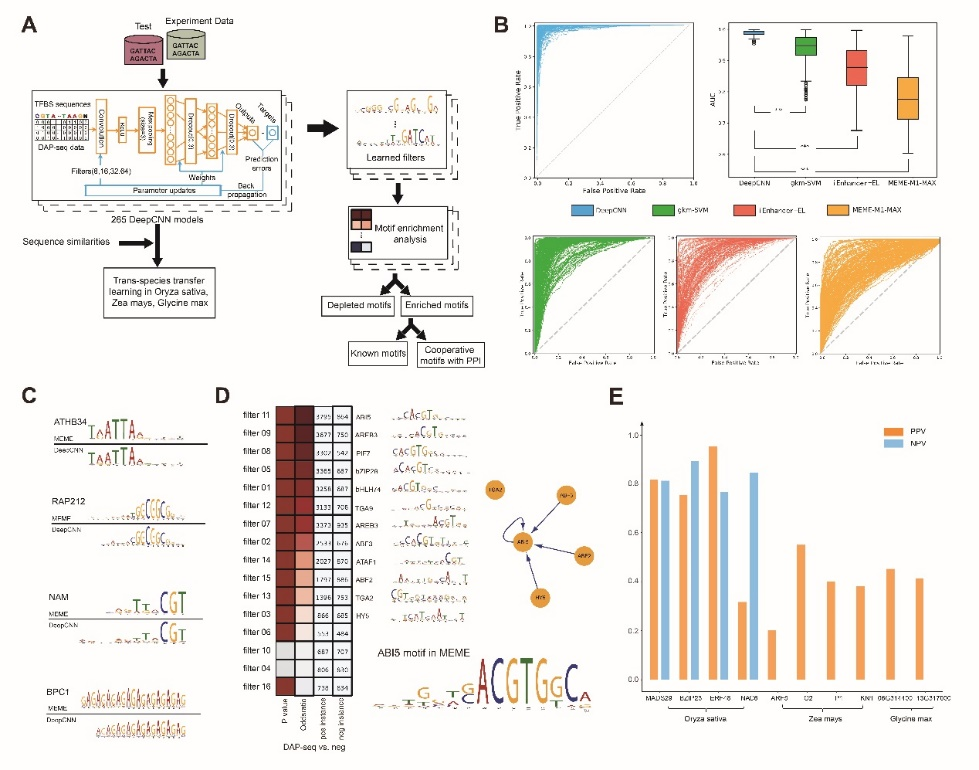
建模结果表明,DeepCNN在265个拟南芥数据集上都获得了很高的预测精确度(平均AUC达0.96),阐明了其在植物TFBS预测方面的可行性。通过进一步深入分析DeepCNN中卷积核的性质,作者提供了模型的生物学可解释性:DeepCNN不仅能学习到当前转录因子在序列当中的关键结合motif,而且能够学习到与该转录因子共同协作的转录因子的结合motif。
最后当使用迁移学习技术尝试从计算的途径解决目前植物TFBS研究问题的困难时,作者发现在不同的植物种类中,迁移学习的表现具有很大的不同。在水稻的十个TF中的三个都取得了比较好的预测效果,BZIP23 、ERF48和MADS29的 PPV(Positive predictive value)分别为0.752、0.951和0.816。而当迁移到玉米和大豆中时,预测效果均不甚理想。这表明迁移学习在植物的跨物种转录因子结合位点预测问题上具有一定的可行性,但是未来我们仍需设计更加有效的迁移学习策略。
为了提供更方便、更优质的生物信息学服务,课题组为此具有高精确率辨别转录因子结合位点的深度卷积神经网络模型搭建了docker镜像,通过下载该镜像并在本地配置可以实现离线预测植物转录因子结合位点的预测功能
